Журнал «Здоровье ребенка» Том 17, №5, 2022
Вернуться к номеру
Механізми дії цитоплазматичних мікроРНК. Частина 4. Рекрутинг декепінгового комплексу DCP1-DCP2. Механізми остаточної деградації мРНК
Авторы: Абатуров О.Є., Бабич В.Л.
Дніпровський державний медичний університет, м. Дніпро, Україна
Рубрики: Педиатрия/Неонатология
Разделы: Справочник специалиста
Версия для печати
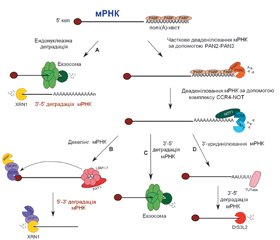
У науковому огляді наведено механізми дії цитоплазматичних мікроРНК, а саме посттранскрипційний сайленсинг: рекрутинг декепінгового комплексу DCP1-DCP2 і порушення взаємодії мРНК з рибосомами. Для написання статті здійснювався пошук інформації з використанням баз даних Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka. Автори показують, що ключовим процесом, який визначає як стабільність мРНК, так і ефективність експресії, є видалення 5’-кінцевого кепа. Декепінг мРНК контролюється кількома прямими й непрямими регуляторами. Комплекс DCP1-DCP2 може рекрутувати безпосередньо на мРНК і опосередковано за допомогою декількох енхансерів декепінгу: PAT1, що безпосередньо взаємодіє з DCP1 і стимулятором декепування; EDC, DDX6. Відомо, що протеїн DCP2 (Nudt20) — це представник консервативної субродини гідролаз Nudix, які каталізують гідроліз невеликих нуклеотидних субстратів. Показано, що протеїн DCP1 — це невелика молекула, яка містить домен EVH1 (enabled/vasodilator-stimulated phosphoprotein homology 1), який зазвичай виконує роль модуля протеїн-протеїнових взаємодій, і C-термінальний домен тримерізації. Відомо, що комплекс DCP1-DCP2 існує у відкритій і закритій конформації, причому закрита конформація має каталітичну активність. Протеїн DCP2 і його енхансерні й кофакторні партнери накопичуються в P-тільцях. Автори показують, що в P-тільцях 5’-монофосфорильована мРНК остаточно розщеплюється під дією 5’-3’-екзорибонуклеази XRN1. Екзорибонуклеази XRN є життєво необхідними ферментами, делеція генів яких супроводжується внутрішньоембріональним летальним кінцем на тлі різних аномалій розвитку органів і систем. Отже, рекрутинг декепінгового комплексу DCP1-DCP2 і порушення взаємодії мРНК з рибосомами в цитоплазмі клітини є механізмами посттранскрипційного сайленсингу. Стабільність мРНК і ефективність експресії визначає видалення 5’-кінцевого кепа. Припинення трансляції обумовлює мРНК. МікроРНК-опосередкована деградація цієї мРНК може здійснюватися як у 3’-5’-, так і в 5’-3’-напрямках молекули.
This scientific review deals with the mechanisms of action of cytoplasmic microRNAs, namely post-transcriptional silencing: recruitment of the DCP1-DCP2 decapping complex and disruption of the interaction of mRNA with ribosomes. To write the article, information was searched using Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka databases. The authors indicate that the key process that determines both mRNA stability and expression efficiency is the removal of the 5’-terminal cap. Decapping of mRNA is controlled by several direct and indirect regulators. The DCP1-DCP2 complex can be recruited directly to mRNA and indirectly with the help of several decapping enhancers: PAT1 directly interacts with DCP1 and the decapping stimulator; EDC, DDX6. It is known that the protein DCP2 (Nudt20) is a representative of the conserved subfamily of Nudix hydrolases, which catalyze the hydrolysis of small nucleotide substrates. It is presented that the DCP1 protein is a small molecule that contains the EVH1 (enabled/vasodilator-stimulated phosphoprotein homology 1) domain, which usually acts as a protein-protein interaction module, and a C-terminal trimerization domain. It is known that the DCP1-DCP2 complex exists in an open and closed conformation, with the closed conformation having catalytic activity. DCP2 protein and its enhancer and cofactor partners accumulate in P-bodies. The authors indicate that in P-bodies, 5’-monophosphorylated mRNA is finally cleaved under the action of 5’-3’-exoribonuclease XRN1. XRN exoribonucleases are vital enzymes whose gene deletion is accompanied by intraembryonic lethality against the background of various abnormalities in the development of organs and systems. Thus, recruitment of the DCP1-DCP2 decapping complex and disruption of the interaction of mRNA with ribosomes in the cytoplasm of the cell are mechanisms of post-transcriptional silencing. The stability of the mRNA and the efficiency of expression determines the removal of the 5’ end cap. Termination of translation is caused by mRNA. MicroRNA-mediated degradation of this mRNA can be carried out both in the 3’-5’ and 5’-3’ directions of the molecule.
мікроРНК; посттранскрипційний сайленсинг; рекрутинг декепінгового комплексу DCP1-DCP2; мікроРНК-опосередкована деградація мРНК; огляд
microRNA; miRNA; miR; post-transcriptional silencing; recruitment of the DCP1-DCP2 decapping complex; мicroRNA-mediated degradation of mRNA; review
Вступ
Висновки
- Arribas-Layton M., Wu D., Lykke-Andersen J., Song H. Structural and functional control of the eukaryotic mRNA decapping machinery. Biochim. Biophys. Acta. 2013 Jun-Jul. 1829 (6–7). 580-9. doi: 10.1016/j.bbagrm.2012.12.006.
- Chang J.H., Xiang S., Tong L. 5’-3’ exoribonucleases. Nicholson A.W., editor. Ribonucleases. Vol. 26. Heidelberg: Springer, 2011. Р. 167-192.
- Charenton C., Taverniti V., Gaudon-Plesse C. et al. Structure of the active form of Dcp1-Dcp2 decapping enzyme bound to m<sup>7</sup>GDP and its Edc3 activator. Nat. Struct. Mol. Biol. 2016 Nov. 23(11). 982-986. doi: 10.1038/nsmb.3300.
- Chen C.Y., Shyu A.B. Deadenylation and P-bodies. Adv. Exp. Med. Biol. 2013. 768. 183-95. doi: 10.1007/978-1-4614-5107-5_11.
- Chen Y., Boland A., Kuzuoğlu-Öztürk D. et al. A DDX6-CNOT1 complex and W-binding pockets in CNOT9 reveal direct links between miRNA target recognition and silencing. Mol. Cell. 2014 Jun 5. 54(5). 737-50. doi: 10.1016/j.molcel.2014.03.034.
- Chowdhury A., Kalurupalle S., Tharun S. Pat1 contributes to the RNA binding activity of the Lsm1-7-Pat1 complex. RNA. 2014 Sep. 20(9). 1465-75. doi: 10.1261/rna.045252.114.
- Corley M., Burns M.C., Yeo G.W. How RNA-Binding Proteins Interact with RNA: Molecules and Mechanisms. Mol. Cell. 2020 Apr 2. 78(1). 9-29. doi: 10.1016/j.molcel.2020.03.011.
- Fabian M.R., Sonenberg N. The mechanics of miRNA-mediated gene silencing: a look under the hood of miRISC. Nat. Struct. Mol. Biol. 2012 Jun 5. 19(6). 586-93. doi: 10.1038/nsmb.2296.
- Ghidini A., Cléry A., Halloy F., Allain F.H.T., Hall J. RNA-PROTACs: Degraders of RNA-Binding Proteins. Angew. Chem. Int. Ed. Engl. 2021 Feb 8. 60(6). 3163-3169. doi: 10.1002/anie.202012330. Epub 2020 Dec 10. PMID: 33108679; PMCID: PMC7898822.
- Grudzien-Nogalska E., Kiledjian M. New insights into decapping enzymes and selective mRNA decay. Wiley Interdiscip. Rev. RNA. 2017 Jan. 8(1). doi: 10.1002/wrna.1379.
- He F., Wu Ch., Jacobson A. Dcp2 C-terminal cis-binding elements control selective targeting of the decapping enzyme by forming distinct decapping complexes. eLife. 2022. 11. e74410. Doi: 10.7554/eLife.74410.
- Hubstenberger A., Courel M., Bénard M., Souquere S., Ernoult-Lange M., Chouaib R. et al. P-Body Purification Reveals the Condensation of Repressed mRNA Regulons. Mol. Cell. 2017 Oct 5. 68(1). 144-157.e5. doi: 10.1016/j.molcel.2017.09.003. Epub 2017 Sep 28. PMID: 28965817.
- Jonas S., Izaurralde E. Towards a molecular understanding of microRNA-mediated gene silencing. Nat. Rev. Genet. 2015 Jul. 16(7). 421-33. doi: 10.1038/nrg3965.
- Jones C.I., Zabolotskaya M.V., Newbury S.F. The 5’ → 3’ exoribonuclease XRN1/Pacman and its functions in cellular processes and development. Wiley Interdiscip. Rev. RNA. 2012 Jul-Aug. 3(4). 455-68. doi: 10.1002/wrna.1109.
- Łabno A., Tomecki R., Dziembowski A. Cytoplasmic RNA decay pathways — Enzymes and mechanisms. Biochim. Biophys. Acta. 2016 Dec. 1863(12). 3125-3147. doi: 10.1016/j.bbamcr.2016.09.023.
- Li Y., Ho E.S., Gunderson S.I., Kiledjian M. Mutational analysis of a Dcp2-binding element reveals general enhancement of decapping by 5’-end stem-loop structures. Nucleic Acids Res. 2009 Apr. 37(7). 2227-37. doi: 10.1093/nar/gkp087.
- Li Y., Kiledjian M. Regulation of mRNA decapping. Wiley Interdiscip. Rev. RNA. 2010 Sep-Oct. 1(2). 253-65. doi: 10.1002/wrna.15.
- Li Y., Song M.G., Kiledjian M. Transcript-specific decapping and regulated stability by the human Dcp2 decapping protein. Mol. Cell. Biol. 2008 Feb. 28(3). 939-48. Doi: 10.1128/MCB.01727-07.
- Ling S.H., Qamra R., Song H. Structural and functional insights into eukaryotic mRNA decapping. Wiley Interdiscip. Rev. RNA. 2011 Mar-Apr. 2(2). 193-208. doi: 10.1002/wrna.44.
- McLennan A.G. The Nudix hydrolase superfamily. Cell. Mol. Life Sci. 2006 Jan. 63(2). 123-43. Doi: 10.1007/s00018-005-5386-7.
- Moser J.J., Fritzler M.J. Relationship of other cytoplasmic ribonucleoprotein bodies (cRNPB) to GW/P bodies. Adv. Exp. Med. Biol. 2013. 768. 213-42. doi: 10.1007/978-1-4614-5107-5_13.
- Nagarajan V.K., Jones C.I., Newbury S.F., Green P.J. XRN 5’→3’ exoribonucleases: structure, mechanisms and functions. Biochim. Biophys. Acta. 2013 Jun-Jul. 1829(6-7). 590-603. doi: 10.1016/j.bbagrm.2013.03.005.
- Ostareck D.H., Naarmann-de Vries I.S., Ostareck-Lederer A. DDX6 and its orthologs as modulators of cellular and viral RNA expression. Wiley Interdiscip. Rev. RNA. 2014 Sep-Oct. 5(5). 659-78. doi: 10.1002/wrna.1237.
- Paquette D.R., Tibble R.W., Daifuku T.S., Gross J.D. Control of mRNA decapping by autoinhibition. Nucleic Acids Res. 2018 Mar 29. doi: 10.1093/nar/gky233.
- Pashler A.L., Towler B.P., Jones C.I., Newbury S.F. The roles of the exoribonucleases DIS3L2 and XRN1 in human disease. Biochem. Soc. Trans. 2016 Oct 15. 44(5). 1377-1384.
- Peterson F.C., Volkman B.F. Diversity of polyproline recognition by EVH1 domains. Front Biosci (Landmark Ed.). 2009 Jan 1. 14. 833-46. PMID: 19273103.
- Robinson S.R., Oliver A.W., Chevassut T.J., Newbury S.F. The 3’ to 5’ Exoribonuclease DIS3: From Structure and Mechanisms to Biological Functions and Role in Human Disease. Biomolecules. 2015 Jul 17. 5(3). 1515-39. doi: 10.3390/biom5031515.
- Schoenberg D.R. Mechanisms of endonuclease-mediated mRNA decay. Wiley Interdiscip. Rev. RNA. 2011 Jul-Aug. 2(4). 582-600. doi: 10.1002/wrna.78.
- She M., Decker C.J., Svergun D.I. et al. Structural basis of dcp2 recognition and activation by dcp1. Mol. Cell. 2008 Feb 15. 29(3). 337-49. doi: 10.1016/j.molcel.2008.01.002.
- Swisher K.D., Parker R. Interactions between Upf1 and the decapping factors Edc3 and Pat1 in Saccharomyces cerevisiae. PLoS One. 2011. 6(10). e26547. doi: 10.1371/journal.pone.0026547.
- Tibble R.W., Depaix A., Kowalska J. et al. Biomolecular condensates amplify mRNA decapping by biasing enzyme conformation. Nat. Chem. Biol. 2021. 17. 615-623. https://doi.org/10.1038/s41589-021-00774-x.
- Tritschler F., Braun J.E., Motz C. et al. DCP1 forms asymmetric trimers to assemble into active mRNA decapping complexes in metazoan. Proc. Natl Acad. Sci USA. 2009 Dec 22. 106(51). 21591-6. doi: 10.1073/pnas.0909871106.
- Valkov E., Jonas S., Weichenrieder O. Mille viae in eukaryotic mRNA decapping. Curr. Opin. Struct. Biol. 2017 Dec. 47. 40-51. doi: 10.1016/j.sbi.2017.05.009.
- Zekri L., Kuzuoğlu-Öztürk D., Izaurralde E. GW182 proteins cause PABP dissociation from silenced miRNA targets in the absence of deadenylation. EMBO J. 2013 Apr 3. 32(7). 1052-65. doi: 10.1038/emboj.2013.44.
- Zhai L.T., Xiang S. mRNA quality control at the 5’ end. J. Zhejiang Univ. Sci B. 2014 May. 15(5). 438-43. doi: 10.1631/jzus.B1400070.

/45.jpg)
/46.jpg)
/47.jpg)